基因表达受多个调控因子在不同水平上的调控,其中包括染色体修饰、DNA甲基化、转录调控、mRNA的可变剪接及降解、转录后调控以及蛋白质修饰等。转录因子(transcription factor,TF)和microRNA (miRNA)作为2种关键的调控因子在转录和转录后水平调控中发挥着至关重要的作用。以TF和miRNA为核心形成的调控网络已经被证实是一种有效的分析与研究生物调控复杂性的方法[1-2]。研究TF与miRNA组成的调控网络将对疾病的发生、发病机理在系统层次上提供重要的线索。尽管目前国内外已经有部分关于TF与miRNA的调控网络研究工作,但关于TF与miRNA调控在研究方法、研究思路尤其是网络模块构建上的研究综述并不多,为了更好地让相关研究人员了解本领域的最新前沿进展,文中将就近年来关于转录因子与miRNA调控网络的研究进行阐述,以期为今后的相关研究提供一定的参考依据。
1 转录因子与microRNA在转录水平,TF已被公认为是重要的调控因子之一,TF通过与位于靶基因启动子区的DNA序列结合形成顺势调控复合体,在转录水平激活或抑制基因的转录[3]。通过活化或抑制下游靶基因,TF可以广泛地调控生物体内的基因表达,并且TF与其靶基因间可以形成复杂的调控网络[4-5]。
MicroRNA (miRNA)是一类长度为18-24nt的非编码小RNA,在进化过程中高度保守,它通过与靶基因的3’UTR区特异性结合从而在转录后水平抑制靶基因的表达或直接降解靶mRNA[6-8]。MiRNA预计调控超过60%的蛋白编码基因并且参与近乎所有已知的细胞进程,例如:细胞周期、分化、细胞增殖、凋亡等[9-11]。近些年来,miRNA参与肿瘤的发生已经成为一个不争的事实,大量的研究也已经证实miRNA具有抑癌和促癌的双重作用,从而为肿瘤的研究又开辟了一条新的思路。
先前研究表明,转录因子可以直接结合在miRNA的启动子区并调控miRNA的表达[12]。这一发现使TF与miRNA 2种处于不同调控水平的网络有机地联系在了一起。全基因组microRNA靶基因预测分析也表明,microRNA的靶点大多都是转录因子[13]。在转录因子与miRNA形成的调控网络中,TF与miRNA在转录调控上共享着同样的调控逻辑[14],TF直接调控miRNA与靶基因,miRNA调控TF与靶基因,一个靶基因可以被多个miRNA所抑制,TF也可同时被TF与miRNA所调控。
2 TF-miRNA调控网络的构建近年来,随着微阵列芯片,ChIP-on-chip和ChIP-seq等高通量实验技术的发展,为研究miRNA与TF之间的调控网络提供了有效途径。尤其是2012年ENCODE[15]项目的完成,为科研工作者提供了90多个细胞系的843个ChIP-seq实验数据(包括组蛋白修饰与对照实验)。这些ChIP-seq实验结果包含了119个转录因子的高分辨率的转录因子结合峰,为今后筛选TF调控miRNA提供了大量的实验数据。
目前构建和分析microRNAs与转录因子调控网络的基本流程主要有以下几个步骤(如图 1所示):1)获取miRNA、基因序列信息,包括已知的转录起始位点(transcription start site,TSS)信息,根据TSS在基因组中的位置,设置假定的启动子区(一般为TSS上游5 kb到下游1 kb)。对于miRNA来说,由于miRNA存在出核后易降解的特性,因此许多miRNA的转录起始位点很难获取,对此情况设定pre-miRNA起始位点的上下游各10 kb作为假定的启动子区。2)通过计算生物学或实验方法获取转录因子结合位点信息。常用的计算生物学方法是通过搜索已知转录因子特征的位置特异性得分矩阵(position-specific scoring matrix,PSSM)来获取未知序列可能存在的转录因子结合位点。但类似的计算生物学预测方法往往存在过高假阳性的问题。由于近来ChIP-seq的广泛应用,通过实验获取某TF的结合位点峰,再通过后期分析得到转录因子结合位点的方法正变为获取转录因子结合位点的主要方法,这种方法优势在于能全面反映活体内真实的转录因子结合DNA情况。3)用获取的转录因子结合位点数据在基因或miRNA的假定启动子区搜索存在匹配,获取TF->miRNA,TF->gene相互作用数据。4)下载gene的3’UTR序列与miRNA的种子序列(seed sequence),通过设定匹配的阈值,在基因的3’UTR搜索可能存在的miRNA结合位点,从而确定miRNA-|gene或miRNA-|TF调控。5)获取蛋白相互作用数据,确定TF-TF,TF-gene,gene-gene间的相互作用关系。6)将第3)、4)、5)步骤产生的数据进行整合,从而构建完整的TF-miRNA-target gene调控网络。利用上述方法人们已经建立了多种TF-miRNA调控网络。例如Reut Shalgi等[13]利用TargetScan和PicTar工具预测人miRNA靶基因,使用TRANSFAC数据库及配套Match TM软件预测保守的TFs结合位点,最后整合累积超几何算法,构建了哺乳动物局部与全局的TF-miRNA调控网络。Cheng等[16]通过整合分析ChIP-seq、RNA-seq以及蛋白质相互作用实验数据,构建了TF-miRNA调控网络,并通过聚类分析、模体分析对网络中的关键网络模块和TF-miRNA的层次关系作了深入的研究。
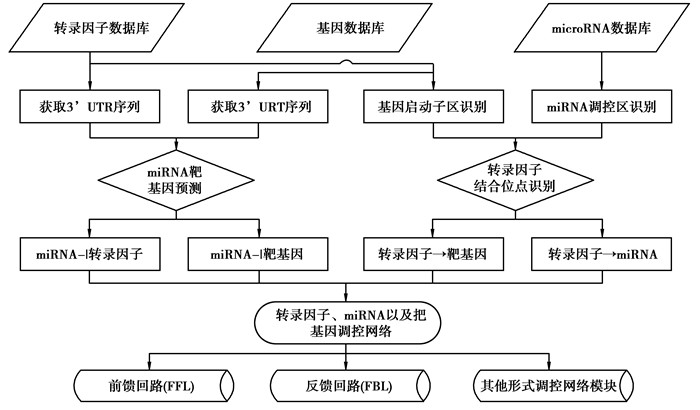
|
图 1 构建和分析microRNAs与转录因子调控网络的基本流程 |
网络模块是指由物理或功能上有联系的节点组成的复合结构,能够行使特定的功能。网络模体则是指在统计学上重复出现的结构模型或更小的子网。目前已经认识到调控网络并不是杂乱无章的,而是具有层次性和整体性。一个复杂的TF-miRNA调控网络是由不同的模块组合而成,而各个模块则是由不同的模体构成[4, 17]。通过探讨模体内miRNA与TF的相互作用方式,能够更好的掌握和理解复杂的TF-miRNA调控网络。在TF-miRNA的调控网络中,最重要的2种网络模体为反馈环和前反馈环。
反馈环由转录因子和miRNA组成,根据作用方式可分为正反馈环和负反馈环,其作用模式为TF与miRNA互相调控。现有的研究发现反馈环在各种生物过程中发挥了重要的作用,特别是在维持体内平衡和调控细胞分化进程中[18]。Aguda等[19]发现由miR-145、Myc及p53构成的负反馈环在协调神经干细胞中的细胞分化和增殖过程起着重要的作用;Yamakuchi等[20]报道miR-34a与p53可形成正反馈环,p53调控miR-34a的转录,同时miR-34a抑制SIRT1 mRNA的翻译。由于SIRT1蛋白下调p53活性,最终p53与miR-34a构成了典型的反馈回路,这一回路因为与肿瘤的疾病发生或干细胞分化存在着非常紧密的联系,因此被广泛地研究[21]。
前反馈回路指转录因子与microRNA协同调控靶基因构成的网络模体,根据类型可分为不连贯的前反馈回路(incoherent FFL)和连贯的前反馈回路(coherent FFL)(如图 2所示)。通过近几年的关于TF-miRNA调控网络中前反馈环的功能研究分析发现其在生物体体内扮演着越来越重要的作用。Angela Re等[22]在人全基因组范围内构建了638个前反馈调控网络,包含2 625个靶基因,101个转录因子和133个miRNA。通过与已有的数据库(miRBase、ECRbase)比较验证了98个TF-miRNA关系对,并利用随机化改组、边缘交换机和完整节点代换分析了与癌症相关的24个TF-miRNA关系对。先前的研究主要倾向于构建典型的3节点FFLs,即由TF,miRNA和基因组成。Sun等[23]对恶性胶质瘤的miRNAs与TF调控网络中的FFLs进行了分析,首次建立了由1个miRNA、1个转录因子和2个共表达的靶基因组成的805个整合的4节点FFLs。通过对3节点的和4节点的FFLs比较分析发现,其所调控的共同靶基因或倾向于在同一生物进程中发挥作用,或定位在相同的细胞组分中,或属于同一蛋白家族。

|
图 2 转录因子、靶基因以及miRNA组成的前反馈回路 |
近年发现,一些miRNA在正常组织与癌组织中表达存在差异,并且多数miRNA在肿瘤中表达下调,大量的研究也已经证实miRNA具有抑癌和促癌的双重作用,从而为肿瘤的研究又开辟了一条新的思路[24]。因此,研究TF-miRNA调控网络将为肿瘤疾病的研究提供重要的理论依据。近来已有一些报道阐明了TF-miRNA调控网络在肿瘤疾病发生中的重要作用。Ye等[25]构建了淋巴母细胞瘤(T-ALL)的TF-miRNA协同调控网络,识别了120个前馈回路,其中miR-19在淋巴母细胞瘤病人与细胞中高度表达,并且与CYLD和NF-κB组成的前馈回路共同影响着下游的信号通路,并参与了淋巴细胞白血病生成的过程。Zeng等[26]利用miRNA序列信息与表达数据构建了转移性与非转移性肝癌的调控网络。通过比较这两类调控网络发现miRNA在转移性肝癌的网络中显示出了更活跃的作用,其中miR-16,miR-30a,let-7e和miR-204在肝癌的转移过程中起了关键的作用。
由于实验条件的限制,目前多数研究还主要停留在通过计算生物学手段构建某一肿瘤疾病的调控网络,然后通过后期的实验验证个别的调控通路的层面。目前还不能从系统水平通过实验研究TF-miRNA调控网络是如何抑制或促进癌症的发生和转移,但随着高通量实验技术的不断出现,通过多个节点甚至网络的层次来研究多个转录因子、miRNA以及基因组成的调控通路在肿瘤疾病发生中所起的作用正逐步变得可行起来。
5 结束语TF与miRNA作为转录水平与转录后水平调控的关键因子,对于细胞的基因表达调控起着重要的作用。因此,两者形成的调控网络的研究对于从系统层面上去了解调控机理与调控通路具有重要的学术意义。过去由于实验条件的限制,往往研究主要集中在单个基因、蛋白或者单条通路的研究上。而随着高通量实验技术的出现与发展,这种研究格局正在被打破。尤其是近几年发展的深度测序技术,为研究人员提供了高质量的、高通量的和多层次的实验数据。尤其是2012年完成的ENCODE计划,通过在70多种细胞系进行的500个ChIP-seq实验获取了超过100个转录因子的结合位点[15]。如何利用这些数据,挖掘其有用的生物学价值并通过实验方法进行验证,是目前摆在每个生命科学工作者面前的重大问题。课题组基于下一代测序数据对Myc与miRNA的前馈回路进行了研究,发现了1 031个与随机产生的FFL存在显著差异的Myc-miRNA前馈回路,这些环路的发现对于更进一步地研究myc与miRNA在肿瘤疾病发生通路中所起的作用提供了一定的理论基础[27]。另外,课题组近期基于ENCODE ChIP-seq数据分析了12个组织的2 347 TF-miRNA相互作用,并构建了相应的调控网络与调控通路数据库,为基于ENCODE的数据挖掘分析提供了新的技术思路与研究方法。
对于TF-miRNA调控网络的研究方面,科研人员还需要不断地探索新的方法、发现新的问题。在数学或计算机等领域合理构建调控网络模型的同时也要符合生物学规律,通过后期的实验验证,有望在多角度、多层次上发现新的调控通路和规律,为学科发展和疾病治疗方面提供有意义的理论和实验依据。
| [1] | Barabasi A L, Oltvai Z N. Network biology:understanding the cell's functional organization[J]. Nature Reviews Genetics, 2004, 5(2): 101–113. DOI:10.1038/nrg1272 |
| [2] | Stites E C, Trampont P C, Ma Z, et al. Network analysis of oncogenic ras activation in cancer[J]. Science, 2007, 318(5849): 463–467. DOI:10.1126/science.1144642 |
| [3] | Vaquerizas J M, Kummerfeld S K, Teichmann S A, et al. A census of human transcription factors:function, expression and evolution[J]. Nature Reviews Genetics, 2009, 10(4): 252–263. DOI:10.1038/nrg2538 |
| [4] | Shen-orr S S, Milo R, Mangan S, et al. Network motifs in the transcriptional regulation network of Escherichia coli[J]. Nature Genetics, 2002, 31(1): 64–68. DOI:10.1038/ng881 |
| [5] | Yu H, Gerstein M. Genomic analysis of the hierarchical structure of regulatory networks[J]. The National Academy Science of the United States of America, 2006, 103(40): 14724–14731. DOI:10.1073/pnas.0508637103 |
| [6] | Saito Y, Suzuki H, Hibi T. The role of microRNAs in gastrointestinal cancers[J]. Journal of Gastroenterol, 2009, 44(19): 18–22. |
| [7] | Baek D, Villen J, Shin C, et al. The impact of microRNAs on protein output[J]. Nature, 2008, 455(7209): 64–71. DOI:10.1038/nature07242 |
| [8] | Farh K K, Grimson A, Jan C, et al. The widespread impact of mammalian MicroRNAs on mRNA repression and evolution[J]. Science, 2005, 310(5755): 1817–1821. DOI:10.1126/science.1121158 |
| [9] | Fabian M R, Sonenberg N, Filipowicz W. Regulation of mRNA translation and stability by microRNAs[J]. Annual Review Biochemistry, 2010, 79: 351–379. DOI:10.1146/annurev-biochem-060308-103103 |
| [10] | Bartel D P. MicroRNAs:genomics, biogenesis, mechanism, and function[J]. Cell, 2004, 116(2): 281–297. DOI:10.1016/S0092-8674(04)00045-5 |
| [11] | Filipowicz W, Bhattacharyya S N, Sonenberg N. Mechanisms of post-transcriptional regulation by microRNAs:are the answers in sight[J]. Nature Reviews Genetics, 2008, 9(2): 102–114. |
| [12] | O'Donnell K A, Wentzel W A, Zeller K I, et al. c-Myc-regulated microRNAs modulate E2F1 expression[J]. Nature, 2005, 435(7043): 839–843. DOI:10.1038/nature03677 |
| [13] | Shalgi R, Lieber D, Oren M, et al. Global and local architecture of the mammalian microRNA-transcription factor regulatory network[J]. PLoS Comput Biol, 2007, 3(7): 131. DOI:10.1371/journal.pcbi.0030131 |
| [14] | Hobert O. Gene regulation by transcription factors and microRNAs[J]. Science, 2008, 319(5871): 1785–1786. DOI:10.1126/science.1151651 |
| [15] | Bernstein B E, Birney E, Dunham I, et al. An integrated encyclopedia of DNA elements in the human genome[J]. Nature, 2012, 489(7414): 57–74. DOI:10.1038/nature11247 |
| [16] | Cheng C, Yan K K, Hwang W, et al. Construction and analysis of an integrated regulatory network derived from high-throughput sequencing data[J]. PLOS Computational Biology, 2011, 7(11): 1–8. |
| [17] | Milo R, Shen-orr S, Itzkovitz S, et al. Network motifs:simple building blocks of complex networks[J]. Science, 2002, 298(5594): 824–827. DOI:10.1126/science.298.5594.824 |
| [18] | Alon U. Network motifs:theory and experimental approaches[J]. Nature Reviews Genetics, 2007, 8(6): 450–461. DOI:10.1038/nrg2102 |
| [19] | Aguda B D, Kim Y, Kim H S, et al. Qualitative network modeling of the Myc-p53 control system of cell proliferation and differentiation[J]. Biophysical Journal, 2011, 101(9): 2082–2091. DOI:10.1016/j.bpj.2011.09.052 |
| [20] | Yamakuchi M, Lowenstein C J. MiR-34, SIRT1 and p53:the feedback loop[J]. Cell Cycle, 2009, 8(5): 712–715. DOI:10.4161/cc.8.5.7753 |
| [21] | Aranha M M, Santos D M, Sola S, et al. MiR-34a regulates mouse neural stem cell differentiation[J]. PLOS One, 2011, 6(8): 1–11. |
| [22] | Re A, Cora D, Taverna D, et al. Genome-wide survey of microRNA-transcription factor feed-forward regulatory circuits in human[J]. Mol Biosyst, 2009, 5(8): 854–867. DOI:10.1039/b900177h |
| [23] | Sun J, Gong X, Purow B, et al. Uncovering microRNA and transcription factor mediated regulatory networks in glioblastoma[J]. PLOS Computational Biology, 2012, 8(7): 1–14. |
| [24] | Barbarotto E, Schmittgen T D, Calin G A. MicroRNAs and cancer:profile, profile, profile[J]. International Journal Cancer, 2008, 122(5): 969–977. |
| [25] | Ye H, Liu X, Lü M, et al. MicroRNA and transcription factor co-regulatory network analysis reveals miR-19 inhibits CYLD in T-cell acute lymphoblastic leukemia[J]. Nucleic Acids Research, 2012, 40(12): 5201–5214. DOI:10.1093/nar/gks175 |
| [26] | Zeng L Y, Yu J, Huang T, et al. Differential combinatorial regulatory network analysis related to venous metastasis of hepatocellular carcinoma[J]. BMC Genomics, 2012, 13(8): 1–14. |
| [27] | Xiong L L, Jiang W, Zhou R, et al. Identification and analysis of the regulatory network of Myc and microRNAs from high-throughput experimental data[J]. Comput Biol Med, 2013, 43(9): 1252–1260. DOI:10.1016/j.compbiomed.2013.06.002 |
 2014, Vol. 37
2014, Vol. 37

