b. 重庆第二师范学院 人事处, 重庆 400067
b. Personnel Department, Chongqing University of Education, Chongqing 400067, P.R.China
癌症是当今医学界所面临的最为严峻的挑战之一[1]。癌症的发生及转移涉及到一系列尚未明确的遗传学和分子生物学过程[2],尤其是近年来越来越多的证据表明组织细胞的信号通路失调是导致癌症的关键因素之一,故酪氨酸蛋白激酶等关键信号蛋白作为抗癌药物的潜在靶标愈发受到重视[3-4]。
由原癌基因SRC编码的c-Src蛋白是非受体型酪氨酸蛋白激酶家族的成员之一,该蛋白的功能异常被认为与多种癌症相关[5-6]。c-Src核心区由一个激酶域CD及两个调节域SH2和SH3构成[7]。传统c-Src蛋白抑制剂的研发主要围绕其激酶域,但多为毒副作用较大的化学药物。同时,c-Src的调节域SH3在c-Src的亚细胞定位、底物识别及介导肿瘤细胞结构形态变化中扮演了重要的角色,有望成为抗癌药物的一个新靶点[8]。由于SH3是典型的肽识别域,专门识别配体的氨基酸片段[9],因此可以设计和多肽分子阻断c-Src的正常功能并抑制肿瘤生长。这一策略的优势在于提高药物靶向特异性,相比于传统化疗最大限度地降低了毒副作用[10]。
鉴于此,众多研究致力于筛选、设计和优化针对c-Src蛋白的多肽类拮抗剂[11]。然而,SH3结构域并非c-Src蛋白所独有。人类蛋白组中迄今已发现300多个SH3结构域[12],其中绝大多数都倾向于识别氨基酸序列中的脯氨酸丰富区域[13]。所以一个多肽配体与c-Src蛋白结合的同时,也有可能与其他蛋白的SH3结构域存在高度的亲和力。所以,候选多肽药物必须与c-Src蛋白特异性结合,从而在实现c-Src靶向抗癌疗效的同时,尽可能避免与其他蛋白的SH3结构域发生混杂性结合,否则有可能干扰与疗效无关的信号通路,引发的不可预期的毒副作用[14]。
本研究围绕多肽与c-Src酪氨酸激酶SH3结构域的特异性结合进行分析,目的在于减少混杂性结合的发生。首先,通过多肽数据库收集已知与c-Src蛋白SH3结构域存在结合关系的多肽作为研究的原型。而后,基于高效的生物信息学工具发现原型多肽氨基酸序列与结合特异性高低之间存在的规律。最后,基于所发现的规律,对相关原型多肽的氨基酸序列进行了优化,使优化后的多肽在结合特异性方面具有显著提升。
1 材料与方法 1.1 搜集原型多肽已知与c-Src蛋白SH3结构域结合的多肽信息采集自Molecular INTeraction (简称MINT) 数据库 (http://mint.bio.uniroma2.it/)。首先,在搜索界面输入c-Src蛋白编码基因的标准名称SRC后,可以从MINT数据库中获得已知与c-Src存在相互作用的分子 (包括蛋白质、化合物和多肽等),笔者仅保留其中的多肽分子。随后,从以上多肽中进一步挑选出具有第一类脯氨酸丰富区域 (class Ⅰ proline-rich motifs) 的多肽分子作进一步分析。第一类脯氨酸丰富区域的特征序列为+xΦPxΦP,其中P代表脯氨酸,x代表任意人类蛋白中常见的氨基酸,Φ代表疏水性氨基酸 (包括丙氨酸,异亮氨酸,亮氨酸,蛋氨酸,苯丙氨酸,脯氨酸,色氨酸,缬胺酸和甘氨酸),+代表带正电荷的氨基酸 (通常为精氨酸或赖胺酸)。
1.2 多肽与各蛋白SH3结构域结合的模拟计算所有原型多肽的氨基酸序列首先被转化为FASTA格式,而后被输入到 (modular domain peptide interaction,MoDPepInt) 在线服务器所提供的SH3PepInt工具进行计算 (http://modpepint.informatik.uni-freiburg.de/SH3PepInt/)。SH3PepInt是一个基于图核方法的SH3结构域与多肽相互作用预测工具。通过使用已知的SH3与多肽之间非线性相互作用数据对支持向量机模型进行训练,SH3PepInt工具能够快捷地预测一条多肽与69个人类蛋白 (其中包括c-Src蛋白) 的SH3结构域是否具有结合潜力。根据SH3PepInt的输出结果,可以评估特定多肽分子除c-Src蛋白以外,还与哪些其他蛋白具有潜在的相互作用关系,从而衡量该多肽与c-Src蛋白结合的特异性。除了c-Src以外,若一个多肽与越多的蛋白发生结合,则代表该多肽的结合特异性越低,即结合混杂性越高。
在SH3PepInt预测结果的基础上,本研究进一步使用CABS-dock在线工具 (http://biocomp.chem.uw.edu.pl/CABSdock) 对特定原型多肽与SH3结构域进行一对一的模拟对接。通过输入原型多肽的氨基酸序列和SH3结构域在Protein Data Bank数据库 (即PDB) 的编号,CABS-dock能够模拟出一系列可能的结合模型。结合模型的准确度以均方根差 (root-mean-square deviation) 为衡量指标,均方根差越低则代表一系列结合模型的整体准确度越高。
2 结果 2.1 原型多肽与SH3结构域结合的混杂性由于SH3结构域主要负责识别富含脯氨酸的氨基酸片段,因此,要研究c-Src蛋白SH3结构域与多肽配体之间的结合,首先需要获得一批具有脯氨酸丰富区域的多肽分子作为研究的原型。为此,笔者首先对MINT数据库[15]进行了检索,从中收集在已发表的实验研究中被证实与c-Src蛋白存在结合关系的多肽。而后,根据第一类脯氨酸丰富区域的氨基酸序列特征,最终确定了10条多肽作为本研究的原型多肽 (详见“1.1搜集原型多肽”)。以这批原型多肽为基础,笔者对多肽氨基酸序列和结合特异性之间的关系进行了探索性研究。
通过传统的生物化学实验来验证多肽分子与靶蛋白之间的结合关系,涉及到多肽合成、蛋白纯化、结合实验等一系列工作,需要投入大量的经费和人力成本,且通常需要较长的研究周期。为了高效率地预测潜在的结合关系,笔者使用了MoDPepInt在线服务器所提供的SH3PepInt工具[16]。该工具仅需要输入目标多肽的氨基酸序列信息,即可根据相关的算法实现多肽分子与靶蛋白结合的模拟计算与预测。在输出结果中,笔者首先发现SH3PepInt工具成功预测了所有原型多肽与c-Src蛋白SH3结构域的结合,充分证明了SH3PepInt预测模型的有效性。在此基础上,笔者进一步分析了SH3PepInt所预测的原型多肽与其他蛋白SH3结构域的相互作用关系。预测结果表明,不同原型多肽在结合特异性上体现出较大的差异 (图 1)。部分原型多肽仅与少数蛋白发生结合,但也有原型多肽体现出较高的混杂性。图 1中矩阵每一行代表一个多肽,每一列代表一个带有SH3识别域的靶蛋白。氨基酸序列中的脯氨酸丰富区域使用涂色标记,多肽与靶蛋白预测存在结合则在矩阵中以涂色标记。
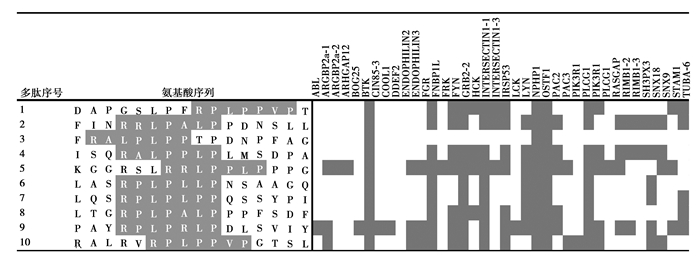
|
图 1 原型多肽的氨基酸序列和预测结合靶点 Figure 1 The amino acid sequences and predicted binding targets of prototype peptides |
鉴于各原型多肽在蛋白结合特异性方面存在较为明显的差异,笔者试图总结出其中的内在规律,尤其是氨基酸序列与结合特异性之间的关联。通过对各原型多肽所结合的蛋白数量进行排序,可以发现第3、第6和第7号原型多肽在SH3PepInt的预测结果中分别仅与3个、9个和10个蛋白的SH3结构域具有潜在的混杂性结合关系,其结合特异性明显优于其他多肽。将以上3条原型多肽与其他多肽进行比对,可以发现第3、第6和第7号原型多肽具有的一个共同特征是脯氨酸丰富区域的第5个氨基酸皆为亮氨酸 (符号为L),而该特征在其他原型多肽中都没有发现 (见图 2)。由此,笔者提出以下假设,脯氨酸丰富区域中的第5个亮氨酸相对有利于c-Src蛋白SH3结构域对多肽的特异性识别,并且降低多肽与其他蛋白的SH3结构域发生混杂性结合的可能性。

|
图 2 原型多肽中脯氨酸丰富区域的第5个氨基酸与结合特异性之间存在一定的关联 Figure 2 The fifth amino acid of proline-rich motif is associated with the binding specificity of prototype peptides |
为了验证以上假设,笔者对除了第3、第6和第7号以外的其他原型多肽进行了优化,将其脯氨酸丰富区域的第5个氨基酸替换为亮氨酸。优化后的原型多肽被重新输入到SH3PepInt工具中与SH3结构域进行模拟结合。结果表明,全部原型多肽在经过优化后不仅保留了对c-Src蛋白的相互作用,而且大多数所结合的其他蛋白数量也有所下降 (见图 3)。笔者将优化后的多肽序列与未优化的原型多肽进行了一对一的比较,并进行了单侧对偶T检验 (见图 4)。结果表明,氨基酸优化后出现的混杂性结合的下降具有统计学意义上的显著性 (P=0.013)。以上数据充分说明,多肽的氨基酸序列与结合特异性之间存在关联,并且这种关联关系能够指导氨基酸序列的优化设计,从而有利于降低候选多肽的毒性风险。
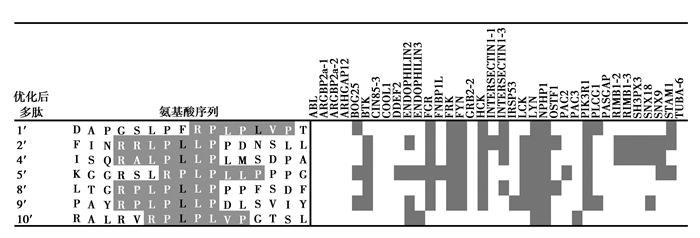
|
图 3 原型多肽经过氨基酸序列优化,将脯氨酸丰富区域的第5个氨基酸替换为亮氨酸后的预测结合靶点 Figure 3 After optimization, thefifth amino acid of proline-rich motif is replaced with Leucine |
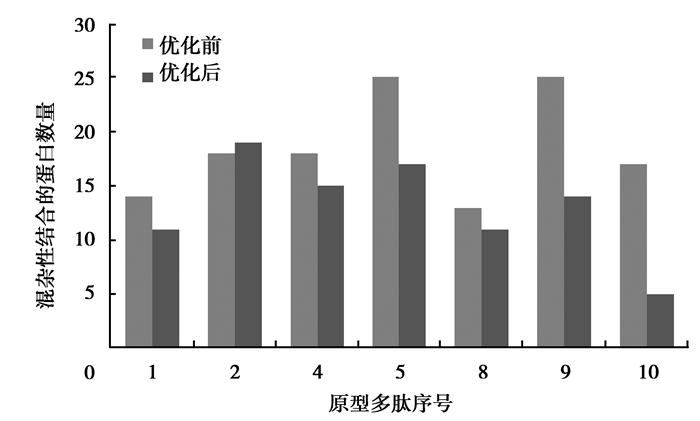
|
图 4 多数原型多肽 (除第2号多肽) 在优化脯氨酸丰富区域的第5个氨基酸后结合特异性得到提升 Figure 4 Most of the prototype peptids, except for No. 2, showed higher binding specificityafter optimization |
为了进一步验证以上结果,笔者使用CABS-dock工具[17]模拟了原型多肽与SH3结构域的具体结合构型。例如,第10号原型多肽经过序列优化后,SH3PepInt工具的预测其不再与HCK蛋白的SH3结构域发生混杂性结合。CABS-dock模拟结果表明,第10号多肽与HCK蛋白的SH3结构域的结合构型在优化前与优化后存在直观的差异 (见图 5(a)、(b))。并且,通过CABS-dock计算的均方根差,可以衡量结合模型的准确性。对比优化前和优化后的10个可能性最高的结合构型,可以发现优化后的结合模型均方根差数值显著高于优化前 (见图 5(c),P=4.64×10-6)。这一差别说明,第10号多肽优化后难以与HCK蛋白形成稳定的结合构型,表现为均方根差的增大,从而进一步验证了氨基酸序列优化提升结合特异性的结论。
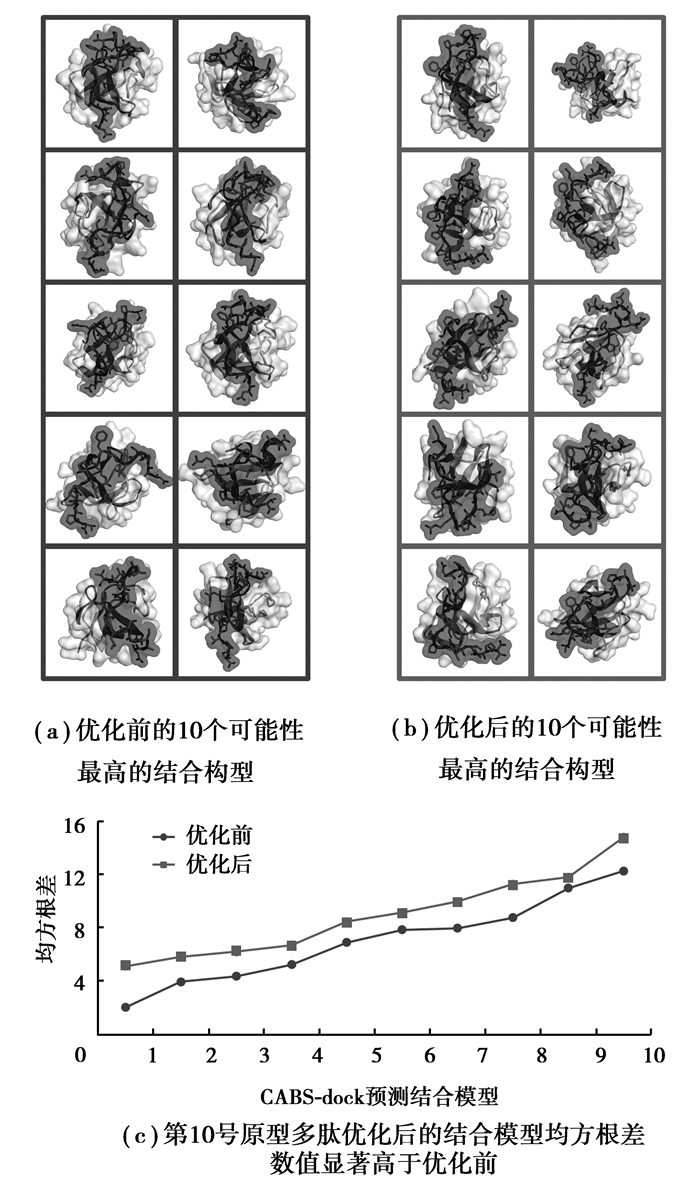
|
图 5 第10号原型多肽与HCK蛋白的SH3结构域的模拟对接结果 Figure 5 The simulated binding between prototype peptide No. 10 and the SH3 domain of HCK protein |
设计多肽拮抗剂使之特异结合c-Src蛋白,且尽可能不与其他蛋白的SH3结构域发生相互作用,一直以来都是一项高难度的研究课题。笔者通过分析多条已知与c-Src的SH3结构域存在结合的原型多肽,发现了多肽序列与蛋白结合特异性之间的规律,从而对原型多肽的氨基酸序列进行一定的优化。计算机模拟的结果表明,优化显著提高了多肽的结合特异性,从而有利于降低毒副作用的风险。
本研究虽然取得了一定的初步结果,但提高多肽与SH3结构域结合的特异性仍需要一系列深入的后续工作。现有的结果主要基于计算机模拟数据,还需要经过生物化学实验的验证,最终确定序列优化在何种程度上提高了多肽与c-Src结合的特异性,从而为设计更安全、毒性更低的多肽类新药提供指导和依据。
| [1] |
邱惠, 张艳, 雷海科, 等.
恶性肿瘤危险因素的Logistic回归分析[J]. 重庆医科大学学报, 2012, 37(3): 251–255.
QIU Hui, ZHANG Yan, LEI Haike, et al. Risk factors related to cancer by Logistic regressive analysis[J]. Journal of Chongqing Medical University, 2012, 37(3): 251–255. (in Chinese) |
| [2] | Koutsogiannouli E, Papavassiliou A, Papanikolaou N. Complexity in cancer biology: is systems biology the answer?[J]. Cancer Medicine, 2013, 2(2): 164–177. DOI:10.1002/cam4.62 |
| [3] | Takeuchi K, Ito F. Receptor tyrosine kinases and targeted cancer therapeutics[J]. Biological & Pharmaceutical Bulletin, 2011, 34(12): 1774–1780. |
| [4] |
李霞.
抗癌药物研究的新进展[J]. 中国药房, 2001, 12(7): 429–431.
LI Xia. The research development ofcancer therapy drug[J]. Journal of Practical Oncology, 2001, 12(7): 429–431. (in Chinese) |
| [5] | Ishizawar R, Parsons S. c-Src and cooperating partners in human cancer[J]. Cancer Cell, 2004, 6(3): 209–214. DOI:10.1016/j.ccr.2004.09.001 |
| [6] | Guarino M. Src signaling in cancer invasion[J]. Journal of Cellular Physiology, 2010, 223(1): 14–26. |
| [7] | Roskoski R. Src protein-tyrosine kinase structure and regulation[J]. Biochemical and Biophysical Research Communications, 2004, 324(4): 1155–1164. DOI:10.1016/j.bbrc.2004.09.171 |
| [8] | Kim L, Song L, Haura E. Src kinases as therapeutic targets for cancer[J]. Nature Reviews Clinical Oncology, 2009, 6(10): 587–595. DOI:10.1038/nrclinonc.2009.129 |
| [9] |
何菁, 夏添松, 王水.
皮动蛋白与肿瘤侵袭性研究进展[J]. 中华肿瘤防治杂志, 2015, 22(1): 72–75.
HE Jing, XIA Tiansong, WANG Shui. Cortactin and tumor invasiveness[J]. Chinese Journal of Cancer Prevention and Treatment, 2015, 22(1): 72–75. (in Chinese) |
| [10] | Craik, Fairlie D, Liras S, et al. The future of peptide-based drugs[J]. Chemical Biology & Drug Design, 2013, 81(1): 136–147. |
| [11] | Sparks A, Quilliam L, Thorn J, et al. Identification and characterization of Src SH3 ligands from phage-displayed random peptide libraries[J]. Journal of Biological Chemistry, 1994, 269(39): 23853–23856. |
| [12] | Kärkkäinen S, Hiipakka M, Wang J, et al. Identification of preferred protein interactions by phage-display of the human Src homology-3 proteome[J]. Embo Reports, 2006, 7(2): 186–191. DOI:10.1038/sj.embor.7400596 |
| [13] | Yu H, Chen J, Feng S, et al. Structural basis for the binding of proline-rich peptides to SH3 domains[J]. Cell, 1994, 76(5): 933–945. DOI:10.1016/0092-8674(94)90367-0 |
| [14] | Rudmann D. On-target and off-target-based toxicologic effects[J]. Toxicologic Pathology, 2013, 41(2): 310–314. DOI:10.1177/0192623312464311 |
| [15] | Licata L, Briganti L, Peluso D, et al. MINT, the molecular interaction database: 2012 update[J]. Nucleic Acids Research, 2012, 40(Database Issue): D857–D861. |
| [16] | Kundu K, Mann M, Costa F, et al. MoDPepInt: an interactive web server for prediction of modular domain-peptide interactions[J]. Bioinformatics, 2014, 30(18): 2668–2669. DOI:10.1093/bioinformatics/btu350 |
| [17] | Kurcinski M, Jamroz M, Blaszczyk M, et al. CABS-dock web server for the flexible docking of peptides to proteins without prior knowledge of the binding site[J]. Nucleic Acids Research, 2015, 43(W1): W419–W424. DOI:10.1093/nar/gkv456 |
 2017, Vol. 40
2017, Vol. 40

